- 카테고리 Genomics > Bacterial Pathogen Analysis
- 수정일2025-11-07 19:11:14
- 레퍼런스
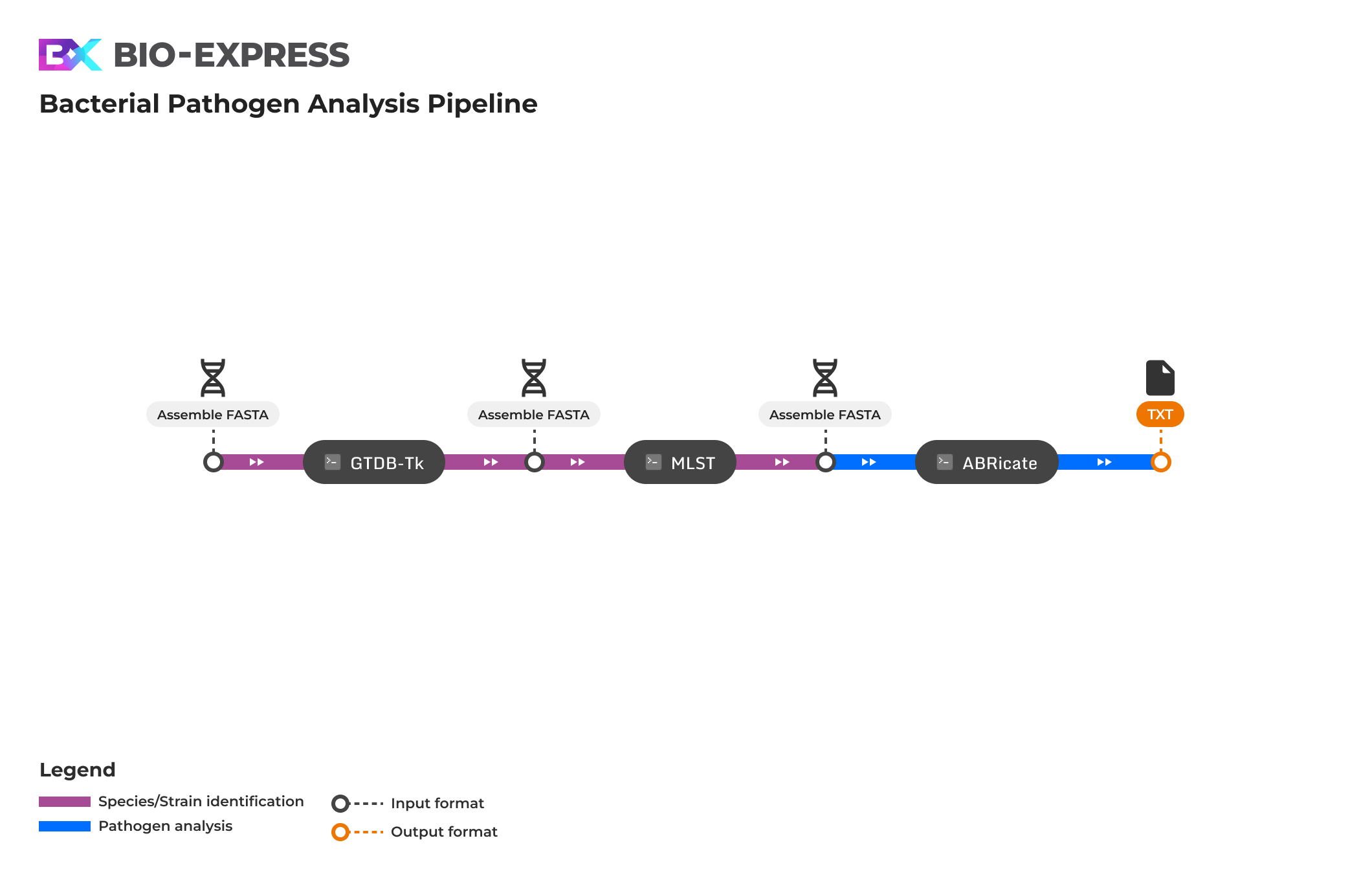
박테리아의 유전체 어셈블리(genome assembly)가 완료된 후에도, 해당 샘플의 정확한 분류학적 위치를 파악하거나 항생제 내성 및 병원성 유전자의 존재 여부를 확인하기 위해서는 따로 후속 분석이 필요합니다. 이 파이프라인은 GTDB-Tk, MLST, ABRicate의 세 가지 도구를 사용해 이러한 기능을 제공하고 있습니다. 세 프로그램 모두 어셈블리가 완료된 유전체 서열 정보를 FASTA 형식의 파일로 입력받아 분석을 시작합니다.
종 동정(Species Identification) 분석은 GTDB-Tk를 사용하여 수행됩니다. 이 도구는 입력된 유전체 서열을 GTDB에 존재하는 방대한 박테리아 서열 정보와 비교 분석하여, 해당 샘플에 대한 종 수준의 분류학적 정보(Taxonomy)를 출력합니다.
이후 MLST 도구를 사용해 종 수준보다 하위 분류군인 균주(Strain) 정보를 확인할 수 있습니다. 해당 도구는 유전체 전체 서열이 아닌, 하우스키핑 유전자(house-keeping gene) 서열만을 활용하여 분류를 수행합니다.
마지막으로 ABRicate를 사용하여 박테리아 유전체 서열 내에 존재하는 항생제 내성 유전자 및 병원성 유전자에 대한 정보를 확인합니다. 이를 위해 CARD, ResFinder, NCBI AMRFinderPlus, VFDB 등 다양한 전문 데이터베이스의 서열 정보를 이용할 수 있습니다.
이러한 단계별 분석을 통해 단순한 서열 정로부터 taxonomy, 임상적 특성을 포괄적으로 파악할 수 있습니다.
파이프라인 모듈
GTDB-Tk
GTDB-Tk(Genome Taxonomy Database Toolkit)는 유전체의 계통 분류(taxonomic classification)를 수행하기 위한 도구로입니다. 완전한 유전체 어셈블리부터 메타게놈으로부터 추출된 MAG(Metagenome-Assembled Genome) 데이터까지 폭넓게 적용할 수 있습니다. GTDB에 포함된 표준화된 계통 분류 체계를 기반으로, 각 유전체의 유전적 거리와 단일복제 마커 유전자의 계통적 위치를 분석하여 분류 계층(계–문–강–목–과–속–종)을 할당합니다. 내부적으로 marker gene alignment, phylogenetic placement, classification 단계를 자동으로 수행하며, RAxML, FastANI 등의 외부 도구를 통합하여 정확하고 재현성 있는 계통 분류를 제공합니다. 이를 통해 다양한 유전체 데이터에 대해 일관되고 재현성 있는 계통 정보를 부여할 수 있습니다.
주요사항
- 이 프로그램은 Genome Taxonomy Database release 226 (R226) 을 기반으로 동작하며, 해당 DB에는 표준화된 세균 및 고세균 참조 유전체가 포함되어 있음.
실행 명령어 예시
GTDB_Tk.sh \ input_dir=./input \ output_dir=./GTDB_Tk/output
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | ./input | 분석에 사용될 binned fasta 파일들이 있는 디렉터리 경로 | |
| Output | Folder | output_dir | ./gtdb_tk/output | 분석 실행 후 결과 파일이 저장될 디텍터리 경로 |
결과
MLST
MLST (Multi-Locus Sequence Typing)는 미생물의 균주(strain)의 타입을 설정하고, 이를 아종 수준에서 분류하는 데 사용되는 도구입니다. 이러한 분석에는 하우스키핑 유전자(housekeeping genes)의 서열 정보가 이용됩니다. 하우스키핑 유전자는 세포 생존에 필수적인 기능을 담당하는 단백질 영역의 정보를 담고 있습니다. 때문에 이 유전자 영역은 매우 보존성이 높고, 같은 종 내의 생물은 거의 동일한 유전자 서열을 가지게 됩니다. 하지만 기능에 영향을 주지 않는 범위 내에서 일부 염기 서열의 변이가 발생할 수 있습니다. MLST는 이 미세한 서열 변이를 이용해 아종을 구분합니다. 우선 서열 변이를 각 유전자 자리(locus) 별로 대립유전자(allele)로 지정합니다. 이 대립유전자의 조합을 서열형(Sequence Type, ST)으로 정의합니다.
실행 명령어 예시
./MLST.sh \ input_dir="./input" \ output_dir="./MLST/output"
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | ./input | 박테리아 게놈 어셈블리 파일이 위치하는 디렉토리 | |
| Output | Folder | output_dir | ./MLST/output | 실행 후 결과물을 저장할 디렉토리 경로 |
결과
ABRicate
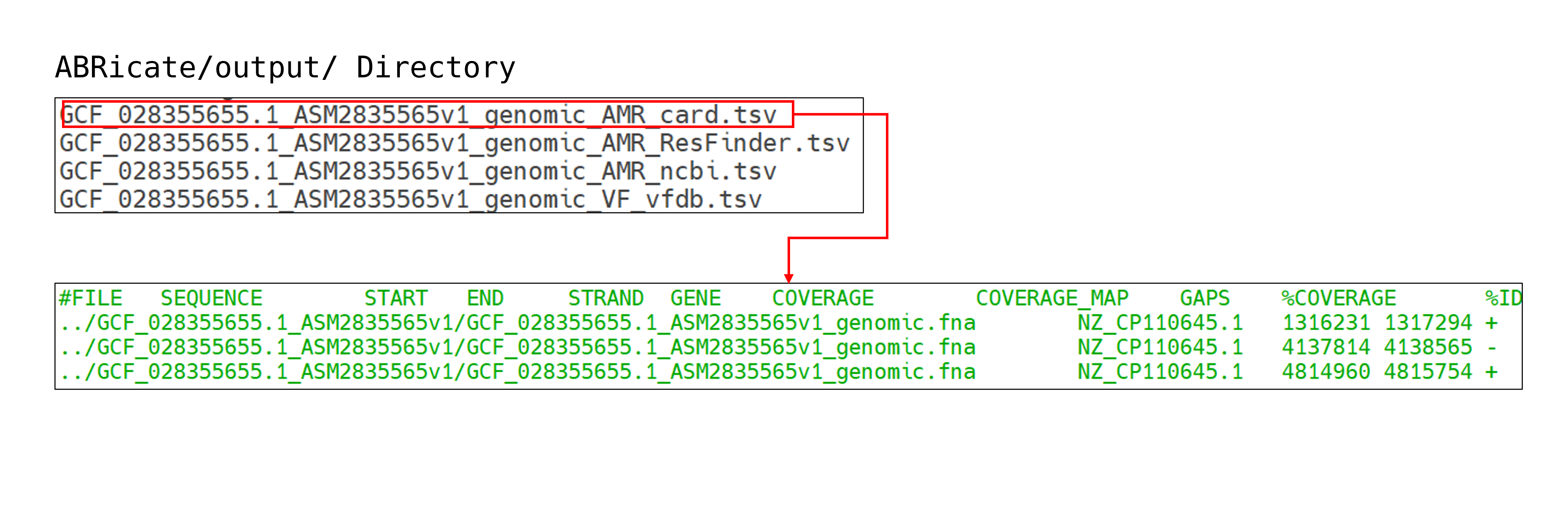
ABRicate는 미생물 유전체학 분야에서 광범위하게 활용되는 도구입니다. 이 도구를 사용하면 미생물 유전체 내에 존재하는 항생제 내성 유전자(Antimicrobial Resistance, AMR) 및 병원성 인자 유전자(Virulence Factor, VF)의 존재 여부를 빠르게 확인할 수 있습니다.
ABRicate는 입력된 박테리아 유전체 서열을 기반으로, 사용자가 선택한 AMR/VF 전문 공공 데이터베이스(DB)에 대해 BLAST 등의 비교 분석을 수행하여 관련 서열의 존재를 확인합니다. 해당 도구에서 선택 가능한 주요 데이터베이스로는 항생제 내성(AMR)과 관련하여 CARD (Comprehensive Antibiotic Resistance Database), ResFinder, ARG-ANNOT, NCBI AMRFinderPlus 등이 있습니다. 병원성 인자(VF)의 경우 VFDB (Virulence Factor Database)를 사용합니다. 그 외에도 플라스미드 서열 정보 확인을 위한 PlasmidFinder, 그리고 E. coli 혈청형 분류에 특화된 EcOH DB 등 다양한 데이터베이스를 지원합니다. ABRicate는 이러한 기능을 통해 미생물의 잠재적 위험성을 평가하는 데 핵심적인 역할을 수행합니다.
실행 명령어 예시
./ABRicate.sh \ input_dir="./input" \ output_dir="./ABricate/output"
실행 스크립트
파라미터
| 옵션 | 유형 | 명칭 | 값 | 설명 | 필수값 |
|---|---|---|---|---|---|
| Input | Folder | input_dir | ./input | 박테리아 게놈 어셈블리 파일이 위치하는 디렉토리 | |
| Output | Folder | output_dir | ./ABricate/output | 실행 후 결과물을 저장할 디렉토리 경로 |